Thème 4: LiverID - Contrôle moléculaire des altérations physiopathologiques de l'identité des cellules hépatiques
Présentation de la thématique de recherche
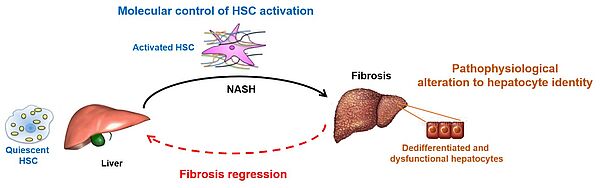
Les maladies du foie constituent un fardeau majeur pour la santé humaine. En particulier, les modes de vie modernes incluant notamment une alimentation et consommation d'alcool excessives sont à l’origine d’un nombre toujours grandissant d'insuffisances hépatiques et de cancers.
Les pathologies du foie découlent d'altérations dans les activités des hépatocytes, le principal type de cellule qui compose cet organe. En effet, un foie dysfonctionnel est caractérisé par des hépatocytes en mauvaise santé, incapables de maintenir des fonctions homéostatiques normales. L'altération des fonctions hépatocytaires s'accompagne de modifications des activités d'autres types de cellules du foie dont les cellules hépatiques stellaires (HSC). En effet, les HSC sont capables d’adopter un phénotype myofibroblastique qui est responsables du développement de la fibrose, un stade avancé des maladies chroniques du foie caractérisé par une altération de la matrice dans laquelle se trouvent les hépatocytes au sein de cet organe.
Notre équipe étudie les mécanismes moléculaires mis en jeu dans les foies malades et qui sous-tendent les altérations des activités des hépatocytes et des HSC. Plus précisément, nous étudions comment les altérations de l'expression des gènes se produisent dans ces cellules et si celles-ci peuvent être corrigées pour rétablir des fonctions cellulaires normales. Enfin, compte tenu du fait que les cellules hépatiques communiquent directement entre elles en échangeant des messages, nous étudions également comment les modifications de l'expression des gènes se traduisent par une modification du dialogue intercellulaire dans les foies malades.
Grâce à nos travaux, nous espérons contribuer à une meilleure compréhension des mécanismes moléculaires qui déclenchent l'altération des fonctions hépatiques dans l'espoir d'identifier de potentielles nouvelles stratégies pour traiter les maladies du foie.
Les travaux récents de notre équipe ont permis de :
- Identifier le sexe et le temps comme principales variables impactant la dérégulation transcriptionnelle dans la stéatohépatite associée à un dysfonctionnement métabolique (MASLD) (Vandel et al. Hepatology 2020; Johanns et al. JHEP Rep 2024).
- Révéler que l'identité des hépatocytes est contrôlée par un large éventail de facteurs de transcription au-delà du réseau de régulation de base précédemment reconnu, y compris le récepteur bêta de l'hormone thyroïdienne (THRB) (Dubois-Chevalier et al. EMBO Rep 2023).
- Identifier le facteur de transcription BNC2 et son O-GlcNacylation comme des pierres angulaires dans l'établissement du programme transcriptionnel myofibroblastique impliqué dans le développement de la fibrose hépatique (Bobowski-Gerard M et al. Nat Commun 2022; Very N et al. Cell Death & Dis 2024).
- Démontrer que des altérations apparemment réversibles des activités métaboliques du foie chez le jeune âge adulte peuvent prédisposer cet organe à un dysfonctionnement exacerbé lorsqu'il est confronté à des désordres métaboliques chroniques plus tard dans la vie (Berthier et al. J Hepatol 2025).
- Caractériser les mécanismes de régulation épigénétique et transcriptionnelle de la transdifférenciation des hépatocytes en cholangiocytes (Vasseur et al. PLoS Biol 2025)
Mots-clés
Bioinformatique ; identité cellulaire ; rythmes circadien ; fibrose ; génomique fonctionnelle ; régulation transcriptionnelle des gènes ; physiopathologie hépatique ; récepteurs nucléaires ; facteurs de transcription
Jérôme EECKHOUTE
Directeur de Recherche 2 CNRS, responsable d'équipe
ORCID : 0000-0002-7222-9264
Jérôme Eeckhoute a obtenu son doctorat en biologie moléculaire à l'Université de Lille en 2003 grâce à des études définissant les conséquences fonctionnelles des mutations du récepteur nucléaire HNF4A associées au diabète (laboratoire de B. Laine). Il a ensuite rejoint le Dana-Farber cancer institute (Harvard Medical School, Boston, USA) où il a participé à la définition de la hiérarchie des facteurs de transcription et de leur interaction avec la chromatine impliquée dans les activités de régulation du récepteur des œstrogènes (laboratoire de Myles Brown). Depuis son recrutement par le CNRS en 2008, il se concentre sur l'application de la génomique fonctionnelle pour déchiffrer les mécanismes moléculaires impliqués dans la (dys)régulation des programmes de régulation transcriptionnelle spécifiques aux cellules/tissus, avec un accent particulier sur les récepteurs nucléaires et la chromatine/épigénomique. Ses intérêts récents portent sur la caractérisation des entités et mécanismes moléculaires responsables de l'établissement et du maintien/de la perte de l'identité cellulaire dans la physiopathologie du foie.
Philippe LEFEBVRE
Directeur de Recherche 1 Inserm, Responsable d’équipe
ORCID : 0000-0002-9366-5129
Philippe Lefebvre a obtenu son doctorat en biochimie à l'ULille1 en 1988, au cours duquel il a étudié le mécanisme d'action des anti-glucocorticoïdes sous la supervision du Pr P. Formstecher. Il a ensuite effectué son stage postdoctoral dans le laboratoire du Dr G.L. Hager au N.I.H., N.C.I., Bethesda pour étudier les mécanismes transcriptionnels régissant l'expression spécifique du type cellulaire du virus de la tumeur mammaire de la souris (MMTV) induite par les glucocorticoïdes et les processus régulés par la chromatine. Depuis 1992, il est à l'INSERM à Lille où il est actuellement directeur de recherche. Il a d'abord créé un groupe de recherche travaillant principalement sur les récepteurs nucléaires de l'acide rétinoïque (RARs, RXRs) et la structure et la régulation des récepteurs orphelins (Nurr1, Nur77) dans la différenciation cellulaire et le cancer. Ses intérêts scientifiques se sont ensuite orientés vers l'étude du rôle des récepteurs nucléaires dans les maladies cardiométaboliques, en se concentrant principalement sur PPARα et FXR. Il dirige maintenant un groupe de recherche dans le laboratoire du Pr Bart Staels et, avec le Dr Jérôme Eeckhoute, se concentre principalement sur les mécanismes moléculaires régulés par les récepteurs nucléaires régissant la physiopathologie du foie, avec un accent particulier sur la NASH et la fibrose.
Lora Autier
Ingénieure d'étude, Université de Lille
Lora Autier a obtenu un Master Biologie-Santé, spécialité oncologie, à l’Université de Lille en 2023. Elle a ensuite rejoint le groupe CER, une entreprise biopharmaceutique spécialisée dans le développement préclinique et située en Belgique, où elle a participé au développement de méthodes analytiques visant à évaluer les réponses immunitaires, la sécurité et l’efficacité de produits biologiques. En septembre 2025, elle a rejoint l’équipe dirigée par le Dr Jérôme Eeckhoute afin d’étudier les mécanismes de régulation transcriptionnelle contrôlant l’identité et la plasticité des cellules hépatiques en contextes physiopathologiques.
Alexandre BERTHIER
Chercheur Post-doctorant
ORCID : 0000-0003-4153-4810
Alexandre Berthier a obtenu son doctorat en sciences de la vie (aspects moléculaires) à l’Université de Besançon en 2008, suite au développement d'outils "biocapteurs/modèles cellulaires" pour l'identification de ligands du récepteur nucléaire aux œstrogènes (ERα) et l'étude des interactions ADN/protéine et protéines/protéines. Il a ensuite étudié la kinase régulée par l’AMP (AMPK) à Grenoble au sein du groupe du Pr. Uwe Schlattner avant de rejoindre en 2010 l’équipe du Dr Philippe Lefebvre à l’Université de Lille. Ses travaux portent sur les récepteurs nucléaires FXR, PPARα et REV-ERBα, et leur rôle dans la régulation circadienne du métabolisme hépatique. Il s'intéresse particulièrement aux mécanismes transcriptionnels impliqués dans la stéatohépatite métabolique (MASH) et la fibrose hépatique, explorant les voies moléculaires qui sous-tendent ces pathologies. Il développe des approches innovantes pour décrypter les interactions moléculaires et les mécanismes métaboliques complexes.
Desiree de BRUIN
Doctorante, Université de Lille
ORCID : 0009-0005-4186-4627
Desiree de Bruin rejoindra l'équipe en février 2025 en tant que doctorante, dans le cadre du réseau doctoral MSCA MIRACLE. Son projet porte sur la plasticité des cellules hépatiques dans l'inflammation et la fibrose liées à la MASH. Elle a une formation en biologie moléculaire (licence) et en sciences médico-légales (master). Elle a travaillé comme technicienne de recherche à l'Institut Hubrecht (Utrecht, Pays-Bas) dans le groupe du prof. Geert Kops, se concentrant sur le marquage endogène des protéines régulatrices du cycle cellulaire en utilisant les techniques CRISPR et l'imagerie des cellules vivantes. Après avoir obtenu sa maîtrise, elle a travaillé en tant qu'associée de recherche au Centre médical universitaire d'Amsterdam (Amsterdam, Pays-Bas) sous la supervision du Dr Peter Henneman, où elle a utilisé les techniques de séquençage de troisième génération pour combiner analyse du génome et de l’épigénome dans un contexte médico-légal.
Julie DUBOIS-CHEVALIER
Ingénieur de Recherche INSERM, Responsable plateforme-bioinformatique
ORCID : 0000-0003-0471-752X
Julie Dubois-Chevalier est spécialisée en bio-informatique et en apprentissage automatique. Elle a obtenu son doctorat en Chimio-informatique à l'Université d'Orléans en 2011, étudiant les concepts de « diversité » et de « représentativité » moléculaires pour proposer une méthode de sélection de molécules pour le criblage à haut débit dans la découverte de médicaments. Elle a rejoint l'équipe en 2012 en tant que post-doctorante sous la direction de Jérôme Eeckhoute pour étudier la coopération des facteurs de transcription dans la génèse de la lignée adipocytaire. Depuis 2016, elle est responsable de la plateforme bioinformatique de l'unité, développant et déployant des outils pour traiter les données omiques. Elle met à profit son expertise en "Machine Learning" pour étudier les mécanismes épigénétiques impliqués dans le contrôle de l'identité des hépatocytes en intégrant des données omiques hétérogènes.
Sandra COURQUET
Ingénieur d’étude Université de Lille
ORCID : 0000-0002-2442-3222
Sandra Courquet a obtenu son master Biologie et Produits de Santé, parcours Relation Hôte-Greffon à l’Université de Besançon en 2017. Elle a ensuite intégré le CNR Echinococcoses/UMR6249 Chrono-Environnement (Besançon), où ses travaux ont principalement porté sur le monitoring du parasite Echinococcus multilocularis, responsable de la maladie hépatique Echinococcose alvéolaire. Puis elle a rejoint le service de recherche clinique d’Oncologie Médicale (CHU de Besançon) en 2022 pour assurer le suivi et la qualité des essais cliniques de phase I à IV. En octobre 2024 elle intègre l’UMR1011 sous la direction du Dr Philippe Lefebvre pour participer à l’étude des mécanismes moléculaires mis en jeu dans les pathologies du foie.
Hélène DEHONDT
Ingénieur d’étude Université de Lille
ORCID : 0000-0003-2900-4673
Hélène Dehondt a obtenu son master Génie Cellulaire et Moléculaire en 2000 à l’Université de Lille. Elle est, ensuite, embauchée au CNRS de Strasbourg pour développer une nouvelle technique de discrimination des mutations dans le cancer colorectal et gérer un sous-projet de recherche européen consistant à mettre en évidence la toxicité des produits issus de l’ionisation des acides gras. Fort de cette expérience, elle intègre l’unité UMR1011 en 2001. Ses missions se répartissent essentiellement en 2 axes : la gestion de plateau technique et la gestion de projets de recherche visant à comprendre les mécanismes moléculaires impliqués dans les maladies métaboliques.
Dmitry GALINOUSKY
Chercheur Post-doctorant,
ORCID : 0000-0002-2522-6052
Dmitry Galinousky a de l'expérience en bioinformatique et en traitement de données et est un expert en génétique moléculaire. Il a développé des approches pour prédire les traits phénotypiques sur la base d'informations omiques, en utilisant des données transcriptomiques, protéomiques et morphométriques. Il a également lors de ses travaux examiné l'état du microbiome intestinal des personnes ayant une longue durée de vie. Dimtry a rejoint l'équipe en avril 2024 pour travailler sur un projet de recherche collaboratif avec la société Genfit. Son activité se focalise sur le traitement de données de RNA-seq à l’échelle du tissu ou de cellules uniques.
Céline GHEERAERT
Ingénieur d’étude Université de Lille
ORCID : 0000-0003-4096-6363
Céline Gheeraert a obtenu un Master en Génie cellulaire et moléculaire à l’Université de Lille en 2006. Elle a débuté sa carrière chez GENFIT, une société de biotechnologie basée à Lille, où elle a contribué à des projets visant à identifier de nouveaux traitements pharmacologiques pour les pathologies liées au syndrome métabolique. En 2008, elle a rejoint l’unité sous la direction du Dr Philippe Lefebvre en tant qu’ingénieure d’études où elle a développé une expertise en techniques d’analyse du cistrome et des modifications épigénomiques (ChIPseq, CUT&RUN, (h)MeDIPseq). Elle a également mis en place et géré un plateau technique spécialisé dans les puces expression (technologie Affymetrix) avant de faire évoluer cette approche technologique vers l’utilisation du séquençage « nouvelle génération » (RNAseq/snRNAseq). Depuis plusieurs années, sous la direction du Dr Jérôme Eeckhoute, elle concentre ses travaux sur l’étude des réseaux transcriptionnels régulant l’identité cellulaire dans la physiopathologie du foie. Ses recherches explorent notamment le rôle des facteurs de transcription dans le maintien et la modulation de l’identité hépatique dans des conditions normales et pathologiques.
Marie Lapage
Ingénieure d'étude, Université de Lille
Marie Lapage est diplômée d’un Master en Biotechnologies, spécialité ingénierie cellulaire et moléculaire, obtenu à l’Université de Lille en 2024. Elle a poursuivi son parcours au sein du laboratoire MIDAC, où elle a mené principalement des travaux portant sur l’évaluation de l’efficacité virucide et/ou bactéricide de produits. En janvier 2026, elle a rejoint l’UMR 1011 sous la direction du Dr Philippe Lefebvre, afin de contribuer à l’étude des mécanismes moléculaires impliqués dans les pathologies hépatiques.
Georgiana TOMA
Chercheur Post-doctorante
Numéro ORCID : 0000-0003-0356-9158
Georgiana Toma a obtenu son doctorat en immunologie médicale à l’Université Martin-Luther de Halle (Saale), en Allemagne. Ses travaux de recherche se situaient à l’interface entre le vieillissement, l’immunologie et les modifications post-traductionnelles des protéines, avec un intérêt particulier pour les lymphocytes T CD8+ et l’acétylation des protéines. Elle a rejoint l’équipe en 2022, en réorientant ses recherches vers les maladies du foie et les rythmes circadiens. Ses travaux actuels portent sur l’étude des interactions entre la glycolyse et les gènes de l’horloge circadienne dans le contexte de la fibrose hépatique.
2026/01 : Bonne année 2026
2025/12 : Félicitations à Ludivine Vasseur pour l'obtention de son doctorat
2025/11 : Deux financements ANR PRC obtenus par l'équipe LiverID!
2025/10 : Note équipe a activement contribué à la fête de la science. Nous avons rencontré des élèves d'écoles primaires et de collèges afin de leur présenter le rôle de l'intelligence artificielle et son lien avec la biologie dans nos travaux de recherches
2025/01 : Céline Gheeraert accède au grade d'ingénieure d'études hors classe (Université de Lille) ! Félicitations Céline pour cette promotion pleinement méritée !
L'équipe 4 de l'UMR1011 a développé de nombreuses expertises :
- Dans les analyses multi-omiques de la régulation transcriptionnelle des gènes (Bulk and scRNAseq, ChIPseq, RIME, Etc)
- Dans les analyses bioinformatiques : analyses de données et apprentissage automatique pour l'intégration de données
- Dans la modélisation in vivo et ex vivo des maladies hépatiques (Cellules primaires de foie murins, modèles murins de maladies du foie, cultures ex vivo de tranches de foies (PCLS))
Liste des diffèrentes publications majeures de l'équipe 4 :
Derepression of the epithelial transcription factor GRHL2 promotes direct hepatocyte-to-cholangiocyte transdifferentiation.
Vasseur L, Gheeraert C, Dubois-Chevalier J, Very N, Guille L, Bou Saleh M, Boulet C, Sobolewski C, Loyer P, Berthier A, Legrand N, Corlu A, Gnemmi V, Lasailly G, Leteurtre E, Galinousky D, Bongiovanni A, Taront S, Toft NI, Grøntved L, Tulasne D, Furlan A, Ntandja-Wandji LC, Staels B, Lefebvre P, Dubuquoy L, Eeckhoute J. PLoS Biol. 2025 Dec 12;23(12):e3003547. doi: 10.1371/journal.pbio.3003547
https://journals.plos.org/plosbiology/article?id=10.1371/journal.pbio.3003547
Unveiling the Molecular Legacy of Transient Insulin Resistance: Implications for Hepatic Metabolic Adaptability. Berthier A, Gheeraert C, Vinod M, Johanns M, Guille L, Haas JT, Dubois-Chevalier J, Eeckhoute J, Staels B, Lefebvre P.J Hepatol. 2025 Feb 11:S0168-8278(25)00080-7.
https://www.journal-of-hepatology.eu/article/S0168-8278(25)00080-7/fulltext
Increased O-GlcNAcylation connects metabolic to transcriptional reprogramming during pathophysiological cell activation. Very N, Staels B, Eeckhoute J. Trends Cell Biol. 2024 Nov 7:S0962-8924(24)00213-7.
pubmed.ncbi.nlm.nih.gov/39516052/
O-GlcNAcylation controls pro-fibrotic transcriptional regulatory signaling in myofibroblasts. Very N, Boulet C, Gheeraert C, Berthier A, Johanns M, Bou Saleh M, Guille L, Bray F, Strub JM, Bobowski-Gerard M, Zummo FP, Vallez E, Molendi-Coste O, Woitrain E, Cianférani S, Montaigne D, Ntandja-Wandji LC, Dubuquoy L, Dubois-Chevalier J, Staels B, Lefebvre P, Eeckhoute J.Cell Death Dis. 2024 Jun 3;15(6):391.
www.nature.com/articles/s41419-024-06773-9
Nuclear receptors: pathophysiological mechanisms and drug targets in liver disease. Dubois V, Lefebvre P, Staels B, Eeckhoute J. Gut 2024 Jun 11:gutjnl-2023-331741.
gut.bmj.com/content/early/2024/06/11/gutjnl-2023-331741.long
The Molecular Circadian Clock Is a Target of Anti-cancer Translation Inhibitors. Berthier A, Gheeraert C, Johanns M, Vinod M, Staels B, Eeckhoute J, Lefebvre P. J Biol Rhythms. 2024 Feb;39(1):20-34.
Time-of-day-dependent variation of the human liver transcriptome and metabolome is disrupted in MASLD. Johanns M, Haas JT, Raverdy V, Vandel J, Chevalier-Dubois J, Guille L, Derudas B, Legendre B, Caiazzo R, Verkindt H, Gnemmi V, Leteurtre E, Derhourhi M, Bonnefond A, Froguel P, Eeckhoute J, Lassailly G, Mathurin P, Pattou F, Staels B, Lefebvre P. JHEP Rep. 2023 Oct 27;6(1):100948.
www.jhep-reports.eu/article/S2589-5559(23)00279-3/fulltext
An extended transcription factor regulatory network controls hepatocyte identity. Dubois-Chevalier J, Gheeraert C, Berthier A, Boulet C, Dubois V, Guille L, Fourcot M, Marot G, Gauthier K, Dubuquoy L, Staels B, Lefebvre P, Eeckhoute J. EMBO rep. 2023 Jul 10:e57020.
www.embopress.org/doi/full/10.15252/embr.202357020
A time- and space-resolved nuclear receptor atlas in mouse liver. Zummo FP, Berthier A, Gheeraert C, Vinod M, Bobowski-Gérard M, Molendi-Coste O, Pineau L, Jung M, Guille L, Chevalier-Dubois J, Dombrowicz D, Staels B, Eeckhoute J, Lefebvre P. J Mol Endocrinol. 2023 Mar 1:JME-23-0017.
https://pubmed.ncbi.nlm.nih.gov/36988391/
Functional genomics uncovers the transcription factor BNC2 as required for myofibroblastic activation in fibrosis. Bobowski-Gerard M, Boulet C, Zummo PF, Dubois-Chevalier J, Gheeraert C, Bou Saleh M, Strub J-M, Farce A, Ploton M, Guille L, Vandel J, Bongiovanni A, Very N, Woitrain E, Deprince A, Lalloyer F, Bauge E, Ferri L, Ntandja-Wandji L-C, Cotte KA, Grangette C, Vallez E, Cianférani S, Raverdy V, Caiazzo R, Gnemmi V, Leteurtre E, Pourcet B, Paumelle R, Ravnskjaer K, Lassailly G, Haas TJ, Mathurin P, Pattou F, Dubuquoy L, Staels B, Lefebvre P, Eeckhoute J.. Nat Commun, 2022 Sep 10;13(1):5324.
www.nature.com/articles/s41467-022-33063-9
Timed use of digoxin prevents heart ischemia–reperfusion injury through a REV-ERBα–UPS signaling pathway. Vinod M, Berthier A, Maréchal X, Gheeraert C, Boutry R, Delhaye S, Annicotte J-S, Duez H, Hovasse A, Cianférani S, Montaigne D, Eeckhoute J, Staels B, Lefebvre P.Nature Cardiovascular Research, 2022
www.nature.com/articles/s44161-022-00148-z
Hepatic molecular signatures highlight the Non-Alcoholic SteatoHepatitis (NASH) sexual dimorphism. Vandel J, Dubois-Chevalier J, Gheeraert C, Derudas B, Raverdy V, Thuillier D, Van Gaal L, Francque S, Pattou F, Staels B, Eeckhoute J, Lefebvre P. Hepatology. 2020 Mar;73(3):920-936. doi: 10.1002/hep.31312.
pubmed.ncbi.nlm.nih.gov/32394476/
The Nuclear Bile Acid Receptor FXR is a PKA- and FOXA2-Sensitive Activator of Fasting Hepatic Gluconeogenesis. Ploton M, Mazuy C, Gheeraert C, Dubois V, Berthier A, Dubois-Chevalier J, Maréchal X, Bantubungi-Blum K, Diemer H, Cianférani S, Strub JM, Helleboid-Chapman A, Eeckhoute J, Staels B, Lefebvre P. J Hepatol. 2018 Jul 4. pii: S0168-8278(18)32175-5.
linkinghub.elsevier.com/retrieve/pii/S0168-8278(18)32175-5
Combinatorial regulation of hepatic cytoplasmic signaling and nuclear transcriptional events by the OGT/REV-ERBα complex. Berthier A, Vinod M, Porez G, Steenackers A, Alexandre J, Yamakawa N, Gheeraert C, Ploton M, Maréchal X, Dubois-Chevalier J, Hovasse A, Schaffer-Reiss C, Cianférani S, Rolando C, Bray F, Duez H, Eeckhoute J, Lefebvre T, Staels B, Lefebvre P. Proc Natl Acad Sci U S A. 2018 Nov 5. pii: 201805397.
pubmed.ncbi.nlm.nih.gov/30397120/
Liste des diffèrents financements obtenus par l'équipe 4 :

La stratégie de notre équipe repose sur l'intégration du travail de recherche expérimental avec l’analyses de données bioinformatiques. Nos projets font ainsi appel au recrutement de personnel contractuel pour travailler avec les membres permanents de l’équipe, en tirant parti de leur expertise complémentaire dans ces domaines.
Nous accueillons des étudiants en Master, des Doctorants et des Post-doctorants intéressés par la gé-nomique fonctionnelle et la physiopathologie du foie. Notre équipe comprend à la fois des biologistes et des bioinformaticiens, ce qui favorise un environnement collaboratif.
Si vous souhaitez nous rejoindre, veuillez contacter :
Dr Jérôme Eeckhoute (jerome.eeckhoute[at]inserm.fr)
Dr Jérôme Eeckhoute
Directeur de recherche CNRS
jerome.eeckhoute[at]inserm.fr
Adresse Postale : Boulevard du Pr Jules Leclercq, Batiment J&K -Faculté de médecine - Pôle Recherche, 59045 Lille Cedex
Dr Philippe Lefebvre
Directeur de recherche INSERM
philippe-claude.lefebvre[at]inserm.fr
Adresse Postale : Boulevard du Pr Jules Leclercq, Batiment J&K -Faculté de médecine - Pôle Recherche, 59045 Lille Cedex
Dubois Vanessa (Post-doctorante) – Professeure d’endocrinologie et cheffe d’équipe, Université de Gand
Boulet Clémence (Ingénieure d’étude)
Bobowski-Gerard Marie (Post-doctorante) – Cheffe de projet R&D, Genfit
Vandel Jimmy (Post-doctorant) – Ingénieur de recherche CNRS
Zummo Francesco (Post-doctorant) – Medical advisor, Novo Nordisk
Guille Loïc (Ingénieur d’étude) – Ingénieur bioinformatique, UC Louvain
Johanns Manuel (Post-doctorant) – Post-doctorant, UC Louvain
Vinod Manjula (Post-doctorante) – Senior scientist, Genfit
Avilkina Viktorija (Post-doctorante) – R&D Project Management, CisBio (Revvity)
Very Ninon (Post-doctorante) – Post-doctorante, Université de Lille
Vasseur Ludivine (Doctorante) – ATER, Université d’Amiens